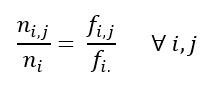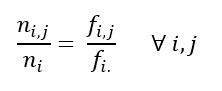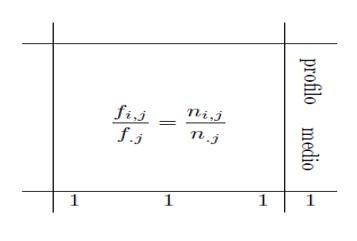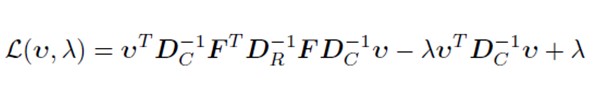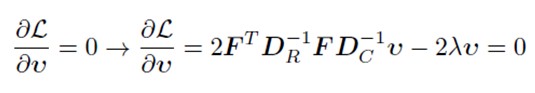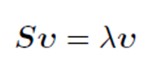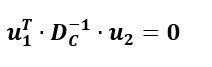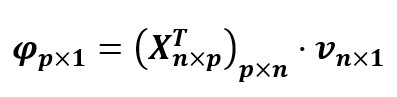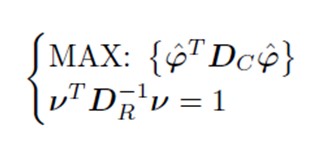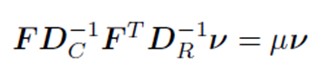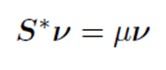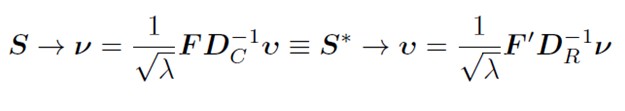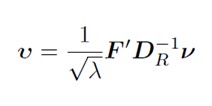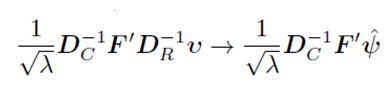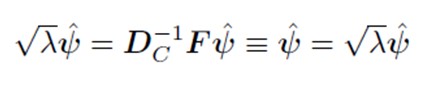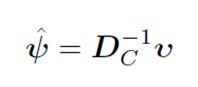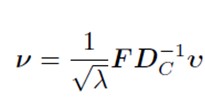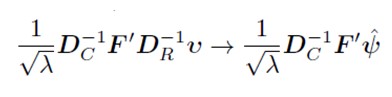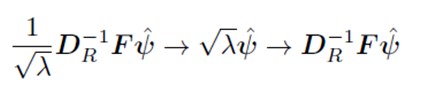Verificarea unei posibile rela?ii între distribu?ia animalelor ?i diferitele regiuni italiene. Datele se refer? la anul 2011, colectate de c?tre b?ncile disponibile pe site-ul Istat.
Ipotez?: diferitele regiuni, în func?ie de caracteristicile teritoriale ?i de nevoile popula?iei, aleg s? creasc? anumite capete de bovine mai degrab? decât altele.
Setul de date:

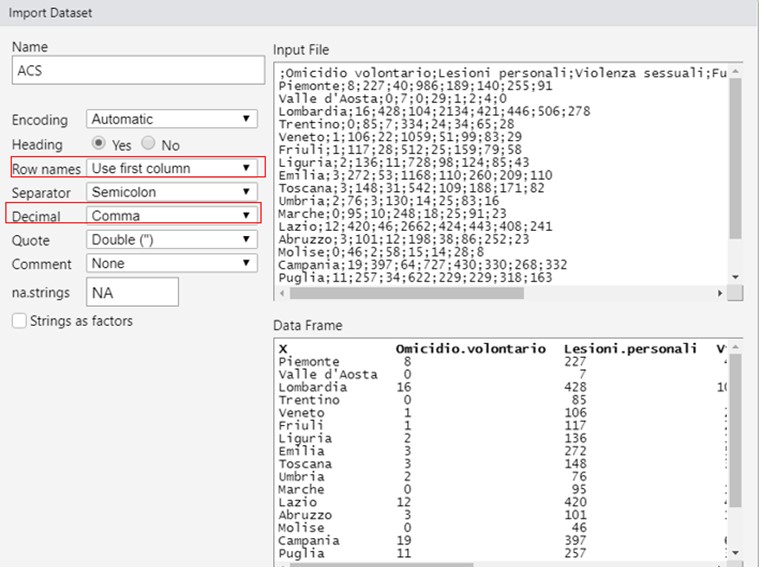
Import?m setul de date:
În câmpul "Row names" (Nume rânduri), selecta?i formularea: "use first column" (utiliza?i prima coloan?) pentru a avea etichetele atât ale indivizilor, cât ?i ale variabilelor pe grafice. În câmpul zecimale, select?m " comma " (virgul?).
With the command:
X<-as.matrix(nome_del_dataset)
Atribuim lui X, ca obiect, setul de date utilizat în analiz?.
Înainte de a putea efectua AC este necesar s? se stabileasc? gradul de interdependen?? dintre cele dou? personaje luate în considerare, aceasta deoarece în cazul în care acestea sunt independente s-ar putea s? nu aib? sens s? se continue AC. Pentru a verifica acest lucru, efectu?m testul chi-p?trat.
chiquadro<-chisq.test(X)
Comanda este urm?toarea:

Se poate observa c? valoarea p este mai mic? decât nivelul de semnifica?ie cel mai frecvent utilizat, ?i anume 0,05. Prin urmare, putem respinge ipoteza nul? de independen?? statistic? între cele dou? variabile ?i putem continua analiza.
Acum dorim s? cre?m o matrice de frecven?e relative F.
Calcul?m num?rul de e?antioane, cu comanda:
n<-sum(X)
?i apoi împ?r?ind matricea de pornire (deci toate frecven?ele comune) la num?rul de e?antioane se ob?ine matricea F. Comand?:
F<-X/n
Urm?torul pas este ob?inerea tabelelor de profiluri de rânduri ?i coloane. Pentru a face acest lucru, în primul rând, este necesar s? se calculeze marginalele rândului ?i ale coloanei. Respectiv, comenzile sunt:
sumrow<-apply(F,1,sum)
sumcol<-apply(F,2,sum)
Apoi calcul?m matricea diagonal? a frecven?elor marginale de rând ?i inversa acesteia cu ajutorul comenzilor:
Dr<-diag(sumrow)
Dr_inv<-solve(Dr)
Acum putem calcula profilurile rândurilor. În termeni matriciali, înmul?im în prealabil inversa matricei diagonale a rândului marginal cu matricea frecven?elor relative. Comanda care trebuie folosit? este:
Pr<-Dr_inv%*%F
Acela?i lucru pentru profilurile coloanelor, ?inând cont de faptul c?, în acest caz, inversa matricei coloanelor trebuie s? fie post-multiplicat? la matricea frecven?elor relative.
Dc<-diag(sumcol)
Dc_inv<-solve(Dc)
Pc<-F%*%Dc_inv
Acum putem calcula distan?ele dintre puncte. Dup? cum am men?ionat deja, exist? dou? tipuri de distan?e: Euclidian? ?i Chi-p?trat.
Profilele rândurilor de distan?e euclidiene sunt:
d_euc_r<-dist(rbind(Pr[1,],Pr[2,]))
Profilele coloanelor de distan?e euclidiene sunt:
d_euc_c<-dist(rbind(Pr[,1],Pr[,2]))
Profilele rândurilor de distan?e chi-p?trat sunt:
d_r<-pr[1,]-pr[2,]
d<-d_r^2/sumcol
d_chi_r<-sqrt(sum(d))
Profilele coloanelor de distan?e chi-p?trat sunt:
dc<-Pr[,1]-Pr[,2]
dc<-dc^2/sumrow
d_chi_c<-sqrt(sum(dc))
Ecua?ia caracteristic? a matricei profilului de rânduri:
S<-t(Pr)%*%Pc
Deoarece matricea S nu este simetric?, este necesar s? o diagonaliz?m pentru a ob?ine S_tilde:
A<-t(F)%*%Dr_inv%*%F #simmetria
Dc_12<-diag(sumcol^(-1/2))
S_tilde<-Dc_12%*%A%*%Dc_12
Acum trebuie s? maximiz?m iner?ia explicat? prin descompunerea matricei în valori proprii ?i vectori proprii:
AC<-eigen(S_tilde)
lambda<-as.matrix(AC$values)
lambda<-lambda[-1,]
w<-AC$vectors
u<-Dc^(1/2)%*%w
u<-u[,-1]
Ecua?ia caracteristic? a matricei profilului de coloane:
S_star<-F%*%Dc_inv%*%t(F)%*%Dr_inv
Pentru a ne deplasa de la u la v, folosim formule de tranzi?ie (deoarece cantitatea de iner?ie explicat? este egal? atât în spa?iul rândurilor, cât ?i în cel al coloanelor).
sq_lambda<-diag((sqrt(lambda))^(-1))
v<-F%*%Dc_inv%*%u%*%sq_lambda
Calcul?m factorii ?i coordonatele, mai întâi spa?iul rândurilor ?i apoi al coloanelor:
fp_r<-Dc_inv%*%u
fp_c<-Dr_inv%*%v
PHI_coord<-Dc_inv%*%t(F)%*%fp_c
PSI_coord<-Dr_inv%*%F%*%fp_r
Se afi?eaz? graficul coordonatelor principale:
PRINCOORD<-rbind(PSI_coord,PHI_coord)
rows<-row.names(X);columns<-colnames(X)
plot(PRINCOORD[,1],PRINCOORD[,2],type="n",
main="Main Coordinates",xlab="Axis1",ylab="Axis2")+ text(PRINCOORD[1:20,1],PRINCOORD[1:20,2],labels=rows,col="springgreen4")
text(PRINCOORD[21:29,1],PRINCOORD[21:29,2],labels=columns,col="violetred")
abline(h=0,v=0,lty=2,lwd=1.5)
Rezult?:
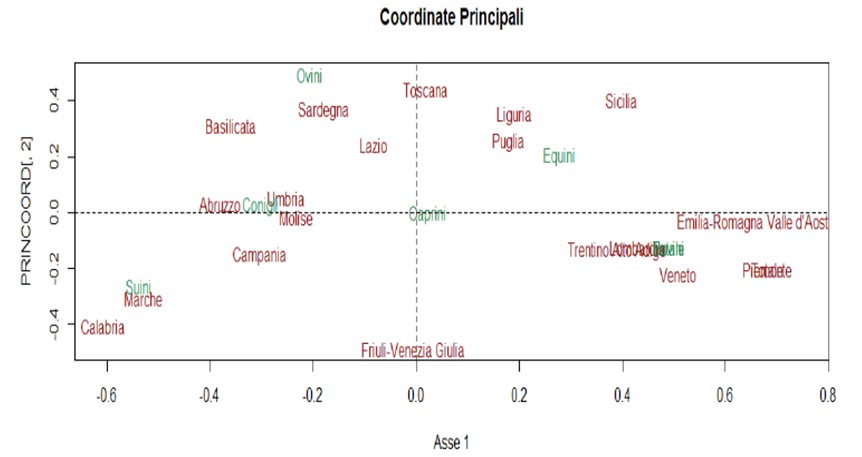
Dac? ne uit?m la acest grafic, putem spune, de exemplu, c? în regiuni precum Abruzzo, Molise, Umbria se cresc în principal iepuri.
Alegem componentele
inertia<-sum(diag(S))-1
sum(lambda)
in_exp<-lambda/inertia
in_exp_<-cumsum(in_exp)
Vizualiz?m rezultatele ob?inute:

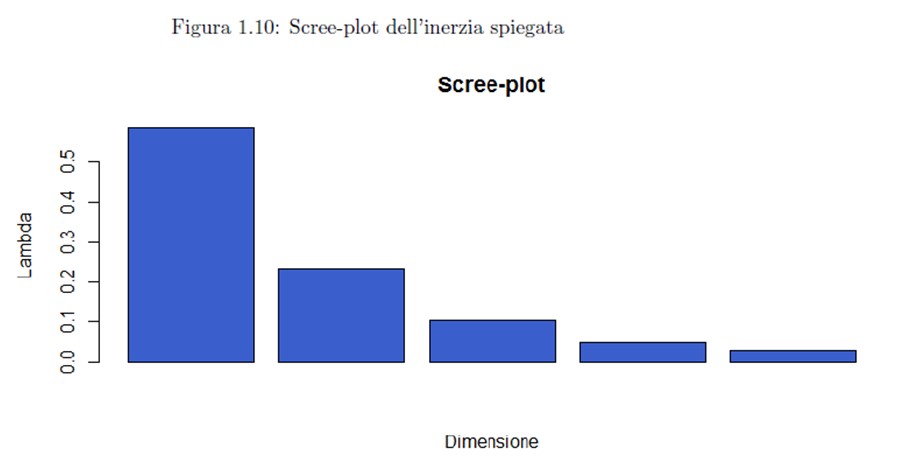
Prima dimensiune explic? singur? 58,57% din variabilitate, iar primele trei împreun? explic? 92,26% din variabilitatea global? a datelor.
Rezultatele ob?inute pot fi afi?ate grafic cu ajutorul graficului scree-plot a iner?iei explicate:
screeplot<-barplot(in_exp,main="Scree-plot inertia", xlab="Size", ylab="Lambda", col="lightblue")
Pentru calitatea reprezent?rii:
- pentru a evalua cât de mult influen?eaz? sau particip? un mod la axa factorial?, se calculeaz? contribu?iile absolute, CA, atât pentru rânduri, cât ?i pentru coloane:
ca_r<-Dr%*%fp_c^2
ca_c<-DC%*%fp_r^2
- Pentru a evalua calitatea reprezent?rii, calcul?m contribu?iile relative, CR. Acestea ofer? o m?sur? mai bun? a reprezent?rii punctelor pe axe ?i este dat? de cosinusul unghiului format de vectorul de proiec?ie al punctului ?i vectorul relativ i (sau j) în punctul i (sau j) din spa?iul s?u original:
G<-matrix(sumcol,20,9,byrow=T)
di<-(Pr-G)^2%*%Dc_inv
d_ig<-apply(di,1,sum)
cos2r<-PSI_coord^2/d_ig
H<-matrix(sumrow,20,9)
dj<-Dr_inv%*%(Pc-H)^2
d_jh<-apply(dj,2,sum)
COS2C<-PHI_coord^2/d_jh
R ofer? un pachet numit FactoMineR pentru analiza coresponden?elor, care adaug? informa?ii despre indivizi ?i variabile ?i v? permite s? crea?i un grafic bidimensional comun al indivizilor ?i variabilelor.
Pe R pentru a putea utiliza acest pachet trebuie mai întâi s? îl desc?rca?i:

Dup? ce l-a?i instalat, trebuie s? îl apela?i cu comanda
library(FactoMineR)
S? trecem la crearea graficului bidimensional Persoane ?i variabile:
CA(X, ncp = 5, row.sup = NULL, col.sup = NULL, quanti.sup=NULL, quali.sup = NULL, graph = TRUE, axes = c(1,2), row.w = NULL)
Grafic, ob?inem:
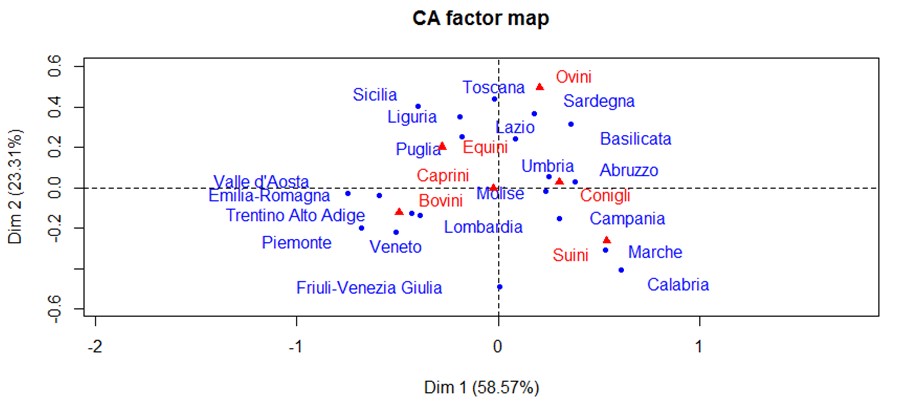
Interpretarea rezultatelor:
Putem spune c? ipoteza ini?ial? este confirmat?. În special, regiunile cele mai dedicate cre?terii ovinelor par a fi Toscana, Sardinia ?i Basilicata, iar acest lucru poate fi explicat prin faptul c? aceste regiuni sunt zone de munte ?i de transhuman??. Caii sunt crescu?i mai ales în Puglia, Liguria ?i Sicilia, deoarece aceste animale au fost întotdeauna folosite pentru munc? în mediul rural. Bovinele sunt prezente în Trentino Alto-Adige, Veneto, Piemont, Lombardia ?i Emilia-Romagna; de fapt, aceste regiuni au o tradi?ie de cre?tere mai dezvoltat? pentru uz alimentar. Iepurii apar mai ales în Umbria, Abruzzo ?i Molise. În schimb, porcii par s? fie crescu?i mai mult în Marche, Campania ?i Molise; Aceste regiuni au, de asemenea, o tradi?ie de cre?tere mai dezvoltat? pentru uz alimentar.
Caprele, pe de alt? parte, sunt plasate la mijlocul axei, probabil pentru c? nu exist? regiuni care s? prefere cre?terea lor.
 Redare audio
Redare audio